Therapieentscheidung und Überwachung bei Immuntherapien
Autorinnen:
Assoc. Prof. Priv.-Doz. Dr. Verena Paulitschke, PhD
Medizinische Universität Wien
Universitätsklinik für Dermatologie
Nina Zila, BSc MSc
FH Campus Wien | University of Applied Sciences
Studiengang Biomedizinische Analytik
Medizinische Universität Wien
Universitätsklinik für Dermatologie
Korrespondenz:
Immuncheckpoint-Inhibitoren (ICIs) revolutionierten die Behandlung des metastasierten Melanoms, doch viele Patient:innen entwickeln Resistenzen. Innovative Biomarker könnten helfen, die Wirksamkeit der Therapie präziser vorherzusagen und Resistenzen frühzeitig zu detektieren. Neben Proteinanalysen ist ein innovativer und vielversprechender Ansatz hierfür die nichtinvasive Analyse von Molekülen aus dem Fingerschweiß, die wertvolle Echtzeitdaten zurTherapietreue und Krankheitsprogression liefern könnte.
Keypoints
-
Robuste Biomarker sind entscheidend: Die Identifizierung prädiktiver Marker ist notwendig, um die Therapieeffektivität von ICIs zu verbessern.
-
Kombination von Markern: Eine synergistische Verwendung von Tumor- und TME-Markern könnte die Prognose und Therapieanpassung erheblich verbessern.
-
Die Fingerschweißanalyse bietet Vorteile: Die innovative und nichtinvasive Methode erlaubt die Überwachung relevanter Metaboliten und könnte zur personalisierten Tumortherapie beitragen.
-
Früherkennung von Therapieresistenzen: Innovative Ansätze könnten es ermöglichen, Therapieresistenzen früher zu identifizieren.
Biomarker zur Überwachung von Immuncheckpoint-Inhibitoren1
Immuncheckpoint-Inhibitoren (ICIs) haben die Behandlung des metastasierten Melanoms stark geprägt. Aktuell sind die 10-Jahres-Überlebensdaten publiziert worden, wobei das mediane Gesamtüberleben bei Patient:innen, die die Anti-PD-1-Immuntherapie erhalten haben, bei 36,9 Monaten liegt.2 Trotzdem entwickeln viele Patient:innen eine Resistenz und sind möglicherweise unnötigen Nebenwirkungen ausgesetzt.3 Um die Wirksamkeit der ICI-Therapie besser vorherzusagen, sind robuste Marker erforderlich.4 Während einige Studien nahelegen, dass PD-1- und PD-L1-Spiegel nützlich sein könnten,5 betrachten andere sie nur als korrelativ, nicht prädiktiv6 – was den Bedarf an aussagekräftigen Markern verdeutlicht.
In der Klinik kommen bereits tumor-basierende Biomarker wie LDH, S100, der Mutationsstatus, besonders der BRAF-Mutationsstatus, in Bezug auf eine „targeted therapy“ oder die immunhistochemische Färbung auf PD-L1 zum Einsatz. Zudem könnten immunbasierte Marker wie Zytokine, die Interferon-gamma-Signatur, Autoantikörper und Immunzellsubtypen sowie das Darmmikrobiom eine Rolle spielen. Dabei könnte eine Kombination von Markern in einem validierten Biomarker-Panel vielversprechend sein.7 Patient:innen mit hohen Eosinophilen- und Lymphozytenzahlen, niedrigem LDH und ohne Metastasen haben ein günstigeres Gesamtüberleben (OS) bei einer ICI-Therapie.8 Diese Kombination von Faktoren zeigt eine stärkere prädiktive Kraft als einzelne Marker. Zudem könnten der entzündliche T-Zell-Tumorphänotyp und der Status von tumorinfiltrierenden Lymphozyten (TILs) als Marker dienen.4
Die Analyse von Immunzellen bei Melanompatient:innen zeigte, dass eine Monozytensignatur vor der Therapie auf ein verlängertes progressionsfreies Überleben (PFS) und OS unter Anti-PD-1-Therapie hinweist.9 Weitere immunsuppressive Mechanismen in der Tumormikroumgebung (TME), wie PD-L2, LAG3 und IDO1, bieten weitere Ansätze für die Immuntherapie.10 Eine Transkriptionssignatur, die mit Angiogenese, Hypoxie und Remodellierung der extrazellulären Matrix (ECM) verbunden ist, wurde als Prädiktor für Resistenz gegen anti-PD-1 identifiziert, wohingegen eine erhöhte Mutationslast und Neoantigene bei Patient:innen mit gutem Therapieansprechen zu finden waren.7 Derzeit wird keiner dieser Marker routinemäßig in der klinischen Praxis verwendet, jedoch könnte eine Kombination von Tumor- und TME-Markern aus Serum- und Tumorgewebe weiterführend sein. Proteine bieten sich für die Analyse besonders gut an, da sie den dynamischen Zustand von Tumor und TME widerspiegeln. In einer aktuellen Studie haben wir diese Proteinanalysen durchgeführt.
Translationale Studie zu prädiktiven Biomarkern1
In einer aktuellen multizentrischen Studie1 führten wir ein Screening von Serum- und Tumorzellen unter Anwendung proteomischer Methoden durch. Hier wurden Serumproben von Melanompatient:innen verwendet, die vor Verabreichung des ersten Zyklus der Anti-PD-1-Immuntherapie gesammelt wurden, um die Daten dann auf Prädiktion ausarbeiten zu können. Zudem wurde eine Vielzahl an klinischen Parametern dokumentiert.
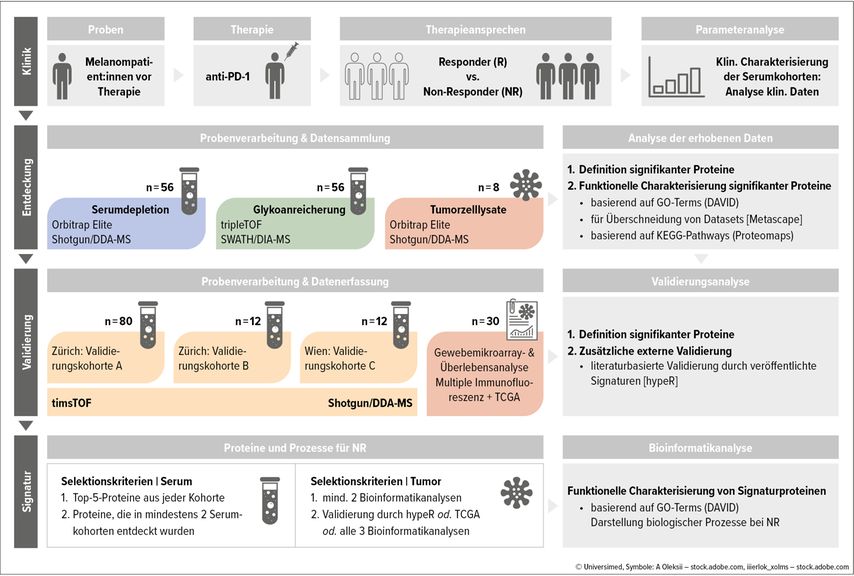
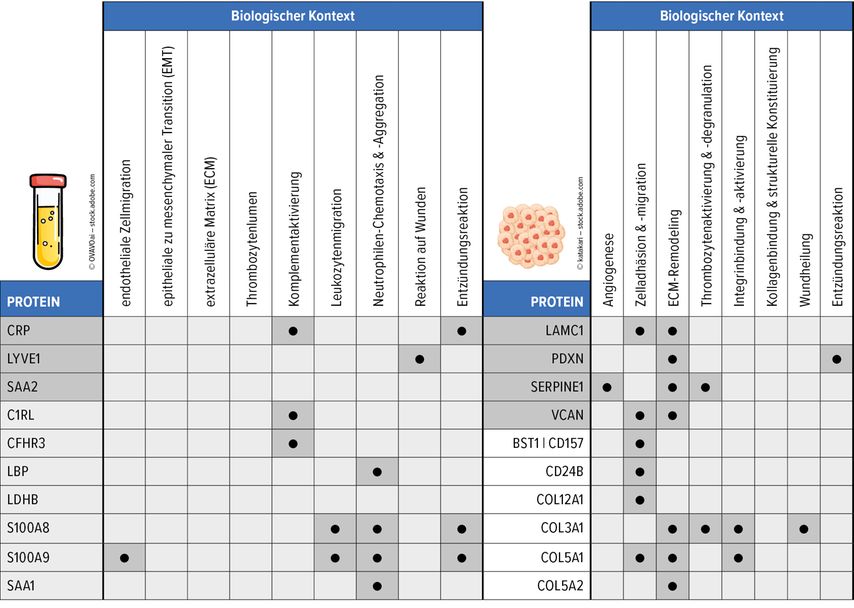
Die Serumkohorte (n=56) wurde mittels Massenspektrometrie (MS) analysiert: Zum einen durch die Abreicherung der zwölf häufigsten Serumproteine und zum anderen durch die Anreicherung glykosylierter Proteine. So konnten Proteine, die im Serum schwer zugänglich sind, besser erkannt werden. Die identifizierten Marker wurden in drei Serumvalidierungskohorten (n=80; n=12; n=17) mit timsTOF-MS analysiert (Abb. 1). Weitere Validierungen basierten auf Literatur und Genomik. Wir analysierten bereits veröffentlichte Signaturen zur Immuntherapie bei Melanomen auf Überschneidungen. Zudem führten wir eine Überlebensanalyse mit Daten des „The Cancer Genome Atlas“ (TCGA) durch, um festzustellen, ob prädiktive Marker auch prognostische Eigenschaften besitzen. Diese Daten wurden integriert, um ein Panel zu erstellen. Zusätzlich wurden Tumormarker durch Immunfluoreszenz an Melanom-Gewebemikroarrays validiert. Durch dieses Vorgehen waren wir in der Lage, eine mögliche prädiktive Signatur herauszuarbeiten, die bei Patient:innen mit schlechtem Therapieansprechen zu finden war (Abb. 2).
Abb. 1: Experimenteller Studienansatz für Baseline-Proben von Melanompatient:innen, die mit anti-PD-1 behandelt wurden. Die primäre Serumkohorte (n=56) wurde mit verschiedenen Techniken analysiert. Basierend auf dem Behandlungserfolg wurden die Patient:innen in Responder (R) und Non-Responder (NR) für nachfolgende bioinformatische Analysen kategorisiert. Schließlich wurden die abgeleiteten Signaturen durch weitere MS-Validierungsanalysen in drei unabhängigen Kohorten sowie durch Literatur- und Überlebensanalysen überprüft. (modifiziert nach Zila N et al. 2024)1
Abb. 2: Markerpanel aus Serum- (li.) und Tumorproben (re.), die durch mehrstufige Analysen zur Vorhersage des Ansprechens auf die PD-1-Blockade bei fortgeschrittenem Melanom bestimmt wurden. Serum-Marker (Top-5-Proteine aus jeder Kohorte und Proteine, die in mindestens 2 Kohorten nachgewiesen wurden) wurden nach strengen Kriterien untersucht. Die 10 Schlüsselmarker aus den 25 Serum-Markern sind als Beispiel dargestellt (dunkelgrau = in 3 Kohorten, hellgrau = in 2 Kohorten). Aufgrund der kleinen Kohorte mussten Tumormarker in mindestens 2 bioinformatischen Analysen identifiziert und durch Literatur oder TCGA-Überlebensanalysen validiert oder in allen 3 bioinformatischen Analysen identifiziert werden. Dies führte zu 23 Tumormarkern, darunter 4 Schlüsselmarker (grau markiert), die sowohl durch Literatur als auch TCGA validiert wurden. (modifiziert nach Zila N et al. 2024)1
Metabolomik
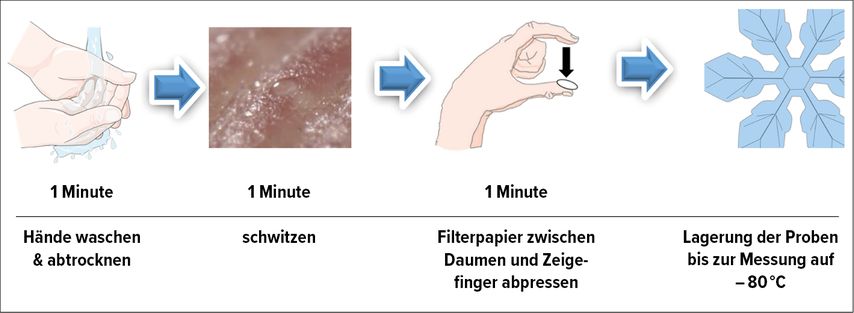
Eine andere innovative und vielversprechende Methode für diagnostische Anwendungen ist die nichtinvasive Schweißanalyse. Es wurde gezeigt, dass Entzündungsmetaboliten11 und Antibiotika12 im Schweiß nachverfolgt werden können. Fingerschweiß, der von den Drüsen der Fingerspitzen abgesondert wird, bietet hierbei besondere Vorteile, da er weniger Variabilität aufweist als Schweiß anderer Körperregionen und klinische Informationen über onkologische Therapeutika liefern könnte.13 Die rasche und nichtinvasive Probengewinnung des Fingerschweißes ist in Abbildung 3 dargestellt. Hiermit konnten beispielsweise bereits Stoffwechselprodukte von antipsychotischen Medikamenten oder Koffein14,15 nachgewiesen werden.
Abb. 3: Ablauf der Fingerschweißgewinnung (zusammengestellt aus Brunmair J et al.15 und https://archive.nerdist.com/a-finger-sweating-looks-far-more-fascinating-up-close-than-youd-imagine )
Ein weiterer Vorteil der Fingerschweißmessung ist die Möglichkeit wiederholter Messungen in kurzen Abständen. Dies erlaubt eine Echtzeitüberwachung wichtiger Faktoren für die Krebsbehandlung, zum Beispiel die therapeutische Medikamentenüberwachung (TDM).16 Fingerschweißanalysen könnten personalisierte Dosierungsstrategien unterstützen und die Therapietreue überwachen. Zudem bietet diese Methode Einblicke in Prozesse wie Entzündungen, die bei der Krebsentwicklung und Therapie eine wichtige Rolle spielen. Damit könnten sich der Immunstatus und die Krankheitsprognose bewerten lassen. Weitere Anwendungsmöglichkeiten liegen in der Früherkennung von Nebenwirkungen (z.B. Immuncheckpoint-Inhibitor-assoziierte interstitielle Entzündung),17 der Bewertung des Ernährungsstatus und der Überwachung psychosozialer Faktoren, die das Wohlbefinden der Patient:innen beeinflussen.
Die Massenspektrometrie des Fingerschweißes mit einer „hands-on time“ von gerade einmal 20 Minuten wurde von der Gruppe von Prof. Gerner an der Analytischen Chemie, Universität Wien, erfolgreich zur Analyse individueller Stoffwechselaktivitäten entwickelt.13,15 Die Vielseitigkeit dieses Ansatzes bietet große Chancen für die personalisierte Patient:innenversorgung.
Ausblick auf die geplante Fingerschweißstudie
In einer ersten Studie mit der Gruppe von Prof. Gerner (Analytische Chemie, Universität Wien) konnten wir zeigen, dass beispielsweise Metaboliten des Tryptophan-Stoffwechsels im Fingerschweiß von Melanompatient:innen hochreguliert sind. Diese Marker wirken immunsuppressiv und könnten daher besonders interessante und relevante Parameter zur Überwachung unter Immuntherapie sein. Darüber hinaus konnten wir zeigen, dass Marker für Dysbiosen im Mikrobiom im Fingerschweiß von Melanompatient:innen erhöht sind. Nachdem bereits eine Korrelation des Mikrobioms mit der Wirkung der Immuntherapie gezeigt werden konnte, könnte das Monitoring dieser Metaboliten unter Immuntherapie ebenfalls von besonderem Interesse sein.
In weiterer Folge möchten wir systematisch Fingerschweißproben vor und während Immuntherapie sammeln. Durch diese umfangreiche Studie könnten wir neue prädiktive und pharmakodynamische Biomarker zur Anti-PD-1-Therapie ermitteln, sowohl auf Protein- wie auch Metabolitenebene. So wäre es möglich, Patient:innen zu stratifizieren, um einerseits keine wertvolle Therapiezeit zu verlieren und/oder auch frühzeitig das Auftreten einer Therapieresistenz zu erkennen. Außerdem erweitern diese Analysen unser Verständnis der ablaufenden Resistenzmechanismen, in die mit neuen Therapieoptionen gezielt eingegriffen werden kann. Durch die Verwendung eines innovativen und nichtinvasiven Ansatzes könnten wir hier eine neue Methode etablieren, die ohne großen Schwellenwert Vorgänge in Patient:innen aufzeichnet und erkennt. Darauf aufbauend könnte die Umsetzung in der Klinik als nächster Schritt gesetzt werden.
Literatur:
1 Zila N et al.: Proteomic profiling of advanced melanoma patients to predict therapeutic response to anti-PD-1 therapy. Clin Cancer Res 2024; 30(1): 159-75 2 Wolchok JD et al.: Final, 10-year outcomes with nivolumab plus ipilimumab in advanced melanoma. N Engl J Med 2024; doi: 10.1056/NEJMoa2407417 (online ahead of print) 3 Hogan SA et al.: Peripheral blood TCR repertoire profiling may facilitate patient stratification for immunotherapy against melanoma. Cancer Immunol Res 2019; 7(1): 77-85 4 Gibney GT et al.: Predictive biomarkers for checkpoint inhibitor-based immunotherapy. Lancet Oncol 2016; 17(12): e542-51 5 Ugurel S et al.: Elevated baseline serum PD-1 or PD-L1 predicts poor outcome of PD-1 inhibition therapy in metastatic melanoma. Ann Oncol 2020; 31: 144-52 6 Strudel M et al.: Melanoma: prognostic factors and factors predictive of response to therapy. Curr Med Chem 2020; 27(17): 2792-813 7 Hugo W et al.: Genomic and transcriptomic features of response to anti-PD-1 therapy in metastatic melanoma. Cell 2017; 168(3): 542 8 Weide B et al.: Baseline biomarkers for outcome of melanoma patients treated with pembrolizumab. Clin Cancer Res 2016; 22(22): 5487-96 9 Krieg C et al.: Author correction: High-dimensional single-cell analysis predicts response to anti-PD-1 immunotherapy. Nat Med 2018; 24(11): 1773-75 10 Zila N et al.: Proteomics-based insights into mitogen-activated protein kinase inhibitor resistance of cerebral melanoma metastases. Clin Proteomics 2018; 15: 13 11 Tu J et al.: A wireless patch for the monitoring of C-reactive protein in sweat. Nat Biomed Eng 2023; 7(10): 1293-306 12 Brasier N et al.: Non-invasive drug monitoring of β-lactam antibiotics using sweat analysis—a pilot study. Front Med (Lausanne) 2020; 7: 476 13 Brunmair J et al.: Finger sweat analysis enables short interval metabolic biomonitoring in humans. Nat Commun 2021; 12(1): 5993 14 Longman K et al.: Noninvasive drug adherence monitoring of antipsychotic patients via finger sweat testing. Front Chem 2023; 11: 1245089 15 Brunmair J et al.: Metabo-tip: a metabolomics platform for lifestyle monitoring supporting the development of novel strategies in predictive, preventive and personalised medicine. EPMA J 2021; 12(2): 141-53 16 Stojanova J et al.: Therapeutic drug monitoring in oncology - what’s out there: a bibliometric evaluation on the topic. Front Oncol 2022; 12: 959741 17Conte P et al.: Drug-induced interstitial lung disease during cancer therapies: expert opinion on diagnosis and treatment. ESMO Open 2022; 7(2): 100404
Das könnte Sie auch interessieren:
Highlights zu Lymphomen
Assoc.Prof. Dr. Thomas Melchardt, PhD zu diesjährigen Highlights des ASCO und EHA im Bereich der Lymphome, darunter die Ergebnisse der Studien SHINE und ECHELON-1
ASH 2020 – Highlights zu den aggressiven Lymphomen
Highlight-Themen der virtuellen ASH-Jahrestagung im Dezember 2020 waren an erster Stelle die Immunonkologika in all ihren Variationen, aber auch Beispiele für innovative Sequenztherapien ...
Aktualisierte Ergebnisse für Blinatumomab bei neu diagnostizierten Patienten
Die Ergebnisse der D-ALBA-Studie bestätigen die Chemotherapie-freie Induktions- und Konsolidierungsstrategie bei erwachsenen Patienten mit Ph+ ALL. Mit einer 3-jährigen ...